抽象
DNA甲基化是许多生物体中重要的表观遗传修饰,可以发生在胞嘧啶或腺嘌呤上。N 6 - 甲基腺嘌呤(6mA)广泛存在于细菌基因组中,在细菌限制修饰系统中起着至关重要的作用。最近,据报道,在从单细胞生物到后生动物的各种真核生物的基因组中存在6mA。关于人类N6amt1(最初被报道为eRF1的谷氨酰胺MTase)是否是推定的6mA DNA MTase存在争议的报道。我们在这里报告了与辅因子SAM复合的人N6amt1-Trm112的晶体结构。结构分析显示Trm112结合N6amt1的疏水表面以稳定其结构,但不直接有助于底物结合和催化。N6amt1的活性位点和潜在的底物结合位点显着带负电荷,因此不适合DNA结合。生化数据证实该复合物不能结合DNA并且对DNA没有MTase活性,但对eRF1的Gln185的甲基化具有活性。我们的结构和生化数据共同证明N6amt1是一种真正的蛋白质MTase而不是DNA MTase。
介绍
DNA甲基化是发生在主要℃的重要修饰5 -胞嘧啶胞嘧啶(5mC),N 4 -胞嘧啶(4mC)和N 6 -腺嘌呤(6毫安)在原核生物和真核生物。这些修改,6毫安最初发现于细菌基因组,主要功能广泛的限制-修饰系统来区分主机和外来病原的DNA,以防止病毒细菌的一部分1,2。然而,这种修饰被认为在后生动物中不存在。最近,随着高通量测序技术和高灵敏度MS方法的发展,据报道在各种真核生物的基因组DNA中也存在6mA,包括真菌,拟南芥,莱茵衣藻,Caenorhabiditis杆线虫,果蝇,斑马鱼,小鼠,猪,和人3,4,5,6,7,8,9,10,11。这些真核生物的基因组上6mA的丰度和分布是完全不同的12。到目前为止,有限的功能性研究表明,DNA6毫安修改是一个潜在的表观遗传标记,并可能在基因转录和染色质重塑的重要作用12,13。
随着在多细胞真核生物的基因组DNA上发现6mA,已经进行了大量努力来鉴定负责写入和去除该修饰的酶。N6amt1(N 6 -腺嘌呤特异性DNA甲基1)是最近建议是第N 6在人细胞中腺嘌呤DNA甲基(转移酶)10。该研究表明,N6amt1的过表达增加了DNA 6mA水平,而该基因的敲低降低了6mA的修饰水平10。此外,重组Flag标记的N6amt1对DNA底物表现出6mA的修饰活性10。此外,原代皮层神经元中N6amt1的体外过表达或敲低也导致6mA水平增加或降低,并且基因组上6mA的积累与消退诱导的基因表达相关14。然而,还有一个矛盾的报告显示,胶质母细胞瘤干细胞中N6amt1的缺失对6mA水平没有影响,纯化的重组N6amt1在体外对DNA底物没有可检测的MTase活性11。因此,关于N6amt1是否是哺乳动物中的真正N 6 -腺嘌呤DNA MTase 仍存在争议。
N6amt1最初被提出作为一个SAM依赖性DNA转移酶,因为它具有的细菌n中的特性NPPY基序6 -腺嘌呤和N 4 -胞嘧啶DNA MTases 15,16。人N6amt1,也称为HemK2,与大肠杆菌 HemK(也称为PrmC)远距离相关。细菌化酶HemK最初提议是一个SAM依赖性DNA转移酶,其直接参与血红素代谢17,18。后来的研究表明,大肠杆菌化酶HemK用作蛋白转移酶可以甲基化多肽释放因子RF1和RF2 19,20。类似地,证明HemK的酵母同源物Mtq2p(也称为YDR140w)催化真核释放因子1(eRF1)21的GGQ基序中谷氨酰胺残基的甲基化。此外,先前的研究还表明N6amt1没有可检测的DNA MTase活性22 ; 相反,它与伴侣蛋白Trm112形成稳定的复合物,并且该复合物在哺乳动物中作为eRF1的谷氨酰胺特异性MTase起作用23。
为了研究N6amt1是否是能够甲基化蛋白质和DNA的双功能酶,我们确定了人类N6amt1-Trm112复合物与辅因子SAM结合的晶体结构,并进行了体外生化分析。我们的结构和生物化学数据一起揭示了N6amt1的活性位点和推定的底物结合位点显着带负电,不适合DNA结合; 该复合物不能与DNA结合,也没有DNA MTase活性; 相反,该复合物对eRF1的Gln185表现出MTase活性。这些结果表明N6amt1是蛋白质MTase而不是DNA MTase。
结果
人N6amt1-Trm112复合物的总体结构
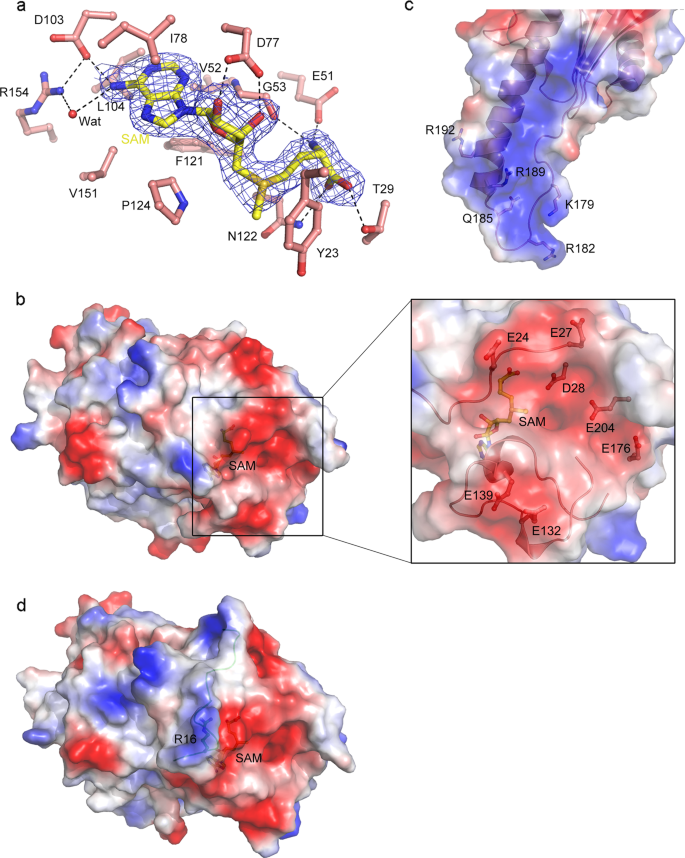
为了研究人N6amt1功能的结构基础,我们从大肠杆菌中表达并纯化了人N6amt1-Trm112复合物。在不存在和存在辅因子S-腺苷 - 甲硫氨酸(SAM)的情况下结晶N6amt1-Trm112复合物均产生SAM结合复合物的晶体,表明SAM可与复合物共纯化。通过SAD方法解析N6amt1-Trm112复合物的结构(表1)。Se-Met衍生蛋白复合物的晶体属于空间群I422,并且在不对称单元中含有两个N6amt1-Trm112分子。天然蛋白质复合物的晶体属于空间群P6 1 22并且在不对称单元中含有一个N6amt1-Trm112分子,并且天然N6amt1-Trm112复合物的结构被精制至2.0Å分辨率(表1)。
在天然N6amt1-Trm112复合物的结构中,除了N末端残基1-18之外,Trm112的所有残基和N6amt1的大多数残基在电子密度中清楚地定义。在Se-Met衍生物N6amt1-Trm112复合物的结构中,尽管结构的分辨率较低,但N6amt1的N末端残基5-18可以构建为具有明显的电子密度(图1a和补充图S 1))。这些结果表明N6amt1的N-末端环(L1环,残基1-27)在溶液中是柔性的。人N6amt1采用典型的I类SAM依赖性MTase折叠,其由一侧夹有三个α-螺旋的中心七链β-折叠板(α1-α3)和另一侧的两个α-螺旋组成(α4)和α5)。除了I类MTases的共有结构核心外,N6amt1还含有两个辅助结构元件:α1螺旋前的柔性L1环和β4链和α4螺旋之间的小插入结构域(图1a)。插入域由三个循环和两个3 10组成螺旋(η2和η3)。活性位点位于MTase结构域的7个β-链的C-末端附近,并且被插入结构域部分覆盖。使用Dali服务器的结构相似性搜索24揭示了人类N6amt1的结构共享具有最高相似性脑炎脑炎微孢子虫(E.脑炎微孢子虫)转移酶和Mtq2的转移酶结构域的大肠杆菌化酶HemK与分别24.2和22.0中,Z分数值(补充表S 1)。有趣的是,E.脑炎微孢子虫 Mtq2甲基化在ERF1的GGQ基序的Gln残基21,和大肠杆菌化酶HemK进行类似的反应来修改RF1和RF2 19,20。这一发现与之前的研究结果一致,表明N6amt1是哺乳动物中eRF1的谷氨酰胺特异性MTase 23。
a带状图中Se-Met衍生物N6amt1-Trm112复合物的总体结构。N6amt1和Trm112分别以鲑鱼和青色青色着色。在衍生复合物结构中定义的N6amt1的额外可见N-末端环以绿色着色。结合的辅助因子SAM用球棍模型显示并着色为黄色。指出了二级结构元件。Trm112的二级结构元素以斜体标记。b Trm112的“S(R / K)CS基序”的结构。与该基序的氢键相互作用中涉及的关键残基用球 - 棒模型显示。氢键用黑色虚线表示。CTrm112和N6amt1在相互作用界面处的静电势表面,这是图1a的开放式视图。表面电荷分布显示为蓝色表示正极,红色表示负极,白色表示中性。为清楚起见,界面分为两个区域以显示详细的交互。d放大窗口显示图1c的两个区域中的详细交互。参与相互作用的残基用球 - 棒模型显示。颜色编码与图1a相同。氢键用黑色虚线表示
伴侣蛋白质人Trm112由四个α-螺旋(α1-α4)形成的螺旋结构域组成,所述α-螺旋包裹由四个反平行β-链(β1-β4)形成的扭曲β-折叠(图1a)。人类Trm112的整体结构与其他MTase-Trm112复合物中的酵母和原生动物Trm112非常相似,但与原生动物Trm112(对于95Cα原子的RMSD为1.8)相似,类似于酵母Trm112(116Cα原子的RMSD)。 )(补充图:S 2和表S = 1)。在所有三种报道的MTase-Trm112复合物中,存在与Trm112结合的Zn 2+,其大约位于β2链的末端。然而,没有与人Trm112结合的金属离子(图1b)。在酵母和原生动物Trm112中,四个Cys残基参与金属离子的结合,这也与周围的残基形成广泛的氢键相互作用,以进一步稳定由N-和C-末端形成的锌结合域。蛋白质(补充图S 2b)。然而,在高等真核生物中,四个Cys残基中的三个被两个Ser残基和一个Arg或Lys残基取代(补充图S 3a)。在人Trm112中,Ser11,Ser103和Cys100在等同位置通过侧链相互作用,并通过主链和侧链与周围残基(Glu102,Ser103,Gly104和Arg105)形成氢键相互作用(图。图1b和补充图:S3a)。此外,Arg14的侧链和主链都与三个保守残基(Glu122,Thr123和Glu124)形成亲水相互作用,这进一步稳定了人Trm112的N-和C-末端。因此,在高等真核生物中,这四种严格或高度保守的残基构成Trm112的非锌结合基序“S(R / K)CS”。
N6amt1和Trm112之间的相互作用
在N6amt1-Trm112复合物中,N6amt1主要通过由N6amt1的β3链和Trm112的β4链形成的平行β-拉链与Trm112相互作用,产生连续的11链β-折叠(图1a)。该复合物的形成掩埋的1200埃的总溶剂可及表面积2。相互作用界面表现出静电和几何互补性(图1c)。N6amt1的残基Ile48,Leu72,Met74,Ala83,Leu87,Pro99,Val100,Ile101,Leu108和Leu112与Trm112的残基Leu4,Leu8,Leu9,Phe41,Met45,Ile113,Pro114和Leu117形成疏水性相互作用,形成界面中心的大疏水区域(图1c,d)。显然,复合物的形成使N6amt1的疏水区域免于暴露于溶剂。这说明有必要共表达N6amt1与Trm112获得稳定的复杂,类似于其他的转移酶,Trm112的复合物25,26,27。
此外,疏水核心被几种亲水相互作用所包围。其中,N6amt1的Ile101的主链胺和羰基分别与Pro114的主链羰基和Trm112的Met116的主链胺形成氢键,这有助于形成平行的β-拉链。界面(图1d,右图)。此外,N6amt1的四个残基(Gln70,Gln98,Gly107和Arg111)与六个Trm112残基(Lys2,Thr5,Asn7,Ser10,Arg44和Lys48)进行七次氢键相互作用。具体地,N6amt1的Gln70的主链与Trm112的Arg44的侧链形成氢键; N6amt1的Gln98侧链与Lys2的主链酰胺和Trm112的Thr5侧链形成两个氢键; N6amt1的Gly107的主链羰基与Trm112的Ser10的主链酰胺形成氢键; N6amt1的Arg111侧链与Asn7和Lys48的主链羰基和Trm112的Asn7侧链形成三个氢键。此外,N6amt1的Glu47和Glu81的侧链分别与Trm112的Lys48和Arg111的侧链形成盐桥(图2)。1d)。序列比对显示,参与复合物形成的大多数残基在两种蛋白质中都是严格或高度保守的(补充图S 3)。
活性位点的结构和潜在的底物结合位点
在N6amt1-Trm112复合物中,在活性位点处存在SAM,具有明确的电子密度(图2a)。SAM与N6amt1的中央β-折叠的C-末端结合并呈现弯曲的构象(图1a),类似于其他I类SAM依赖性MTase 28的结构。SAM的腺嘌呤部分与周围的残基(Val52,Ile78,Leu104,Phe121,Pro124和Val151)形成广泛的疏水相互作用,此外,其N6基团与Asp103的侧链形成直接氢键,并与其间接形成氢键。 Arg154的侧链通过水分子(图2a)。核糖的两个羟基与Asp77的侧链形成氢键。甲硫氨酸部分通过其酰胺基与Glu51的侧链和Gly53的主链羰基相互作用,并通过其羧基与Thr29和Asn122的侧链相互作用。Tyr23的侧链与甲基和SAM的硫原子相邻,可能在甲基化过程中发挥作用。序列比对显示SAM结合中涉及的关键残基在来自不同物种的N6amt1中是严格或高度保守的(补充图S 3b)。
SAM与N6amt1 的交互。与辅因子相互作用的残基用球 - 棒模型显示。参与氢键网络的水分子用球体显示并用红色着色。用蓝色网格显示SAM 的代表性复合模拟退火省略图(2Fo-Fc,轮廓为1.0σ水平)。b与图1a相同视图中的天然N6amt1-Trm112复合物的静电势表面。放大窗口显示组成活性位点的酸性残基和潜在的底物结合位点。CeRF1-eRF3复合物(PDB代码3E1Y)中人eRF1的GGQ基序周围的静电势表面。Gln185和相邻的碱性残基用球棒模型显示。d与图1a相同的视图中的Se-Met衍生物N6amt1-Trm112复合物的静电势表面。额外可见的N端环以带状显示并以绿色着色。辅助因子SAM和关键残基用球棍模型显示
由于关于N6amt1是否是哺乳动物中真正的6mA DNA MTase仍然是一个有争议的问题,我们进行了结构分析以探索N6amt1的底物特异性。结构相似性搜索表明N6amt1股与Mtq2和化酶HemK最高的相似性,以及类似于Mtq2多于化酶HemK(补充表S = 1和图:S 4A)。先前的结构和功能研究表明,到目前为止鉴定的大多数I类MTase含有辅助结构元件,这些辅助结构元件插入整个共有的7-β链折叠并且似乎促进底物结合和识别28。除了MTase结构域,大肠杆菌化酶HemK包含其参与识别和基板RF1(补充图S的结合大的N末端结构域4A)。然而,N6amt1和Mtq2的附件结构元件是相对小的(图1A和补充图š 3B)。与Mtq2类似,N6amt1表现出围绕SAM甲基的相对平坦,开放和带负电的表面,其可能是底物结合位点(图2b和补充图S 4b)。)。这个带负电荷的表面由几个酸性残基组成,包括来自L1环的Glu24和Glu27,来自插入结构域的Glu132和Glu139,以及来自MTase结构域的Asp28,Glu176和Glu204,这可能有助于底物结合和识别(图2b)。显然,这种带负电荷的N6amt1底物结合位点不适合于DNA底物的带负电荷的磷酸骨架的结合。另一方面,报道的人eRF1-eRF3复合物的结构分析显示人eRF1的Gln185被几个碱性残基包围,特别是Lys179,Arg182,Arg189和Arg192形成带正电荷的表面贴片,表明eRF1是合适的。 N6amt1的底物(图2c)。
在天然N6amt1-Trm112复合物的结构中,L1环的N末端是无序的,L1环仅含有残基19-27; 然而,在Se-Met衍生物N6amt1-Trm112复合物的结构中,定义了L1环的残基5-18(图1a)。L1环的额外可见区域由三个基本残基(His11,His13和Arg16)组成,它们形成一个小的带正电的表面贴片,但不会改变潜在的底物结合位点的整体电荷特性(图2d))。考虑到N6amt1的L1环在溶液中相对灵活,是否有助于DNA底物的结合和识别将在后来的生化研究中得到验证。
在N6amt1-Trm112复合物中,Nmam1的大部分疏水表面被Trm112在暴露于溶剂时屏蔽(图1c)。N6amt1只有在与Trm112 23形成复合物时才能有效甲基化人eRF1的Gln185 。除了N6amt1和其同系物的酵母Mtq2,Trm112也可以形成与其它I类MTases,包括Bud23,Trm9和TRM11,其催化不同的基材,包括rRNA和tRNA的甲基化复合物25,26,27,29。在那些MTase-Trm112复合物中,Trm112被认为是MTases 30的伙伴和激活剂。。为了研究Trm112是否参与底物结合和识别N6amt1的作用,我们比较了N6amt1-Trm112复杂与Bud23-Trm112和Trm9-Trm112的报道结构的结构复合物25,26。结构比较显示Bud23和Trm9可以与N6amt1良好叠加,尽管序列同一性低(补充表S1和图4a)。此外,在所有MTase-Trm112复合物中,Trm112以类似方式与MTase相互作用(补充图S 4a))。特别地,Trm112的β4链与MTases的β3链相互作用以形成平行的β-拉链,产生连续的11链β-折叠。此外,在其他MTase-Trm112复合物中也可以发现N6amt1和Trm112之间的几种亲水相互作用。例如,N6amt1的Gln98和LYS2和Trm112的Thr5之间和N6amt1的Arg111和Asn7和Trm112的Lys48之间的亲水性相互作用具有在Bud23-Trm112和Trm9-Trm112络合物对应的相互作用(补充图:S 4C)。这些结果表明Trm112与那些MTase的相互作用模式从酵母到人类是保守的。在所有三种MTase中,活性位点位于MTase结构域的七个β-链的C-末端附近。然而,Bud23和Trm9中活性位点周围的结构表面带正电荷,这适合于RNA底物的结合; 相反,N6amt1中活性位点周围的结构表面带负电,这不适合核苷酸底物的结合(补充图S 4b和图2b))。由于Trm112与这些复合物中MTase的活性位点远离结合,其结合似乎对活性位点和底物结合位点的化学性质和结构没有直接影响,表明Trm112不直接参与底物约束和认可。然而,先前对Mtq2-Trm112和Trm11-Trm112复合物的生化研究表明,位于Trm112表面的几个残基突变远离与MTase的相互作用界面可能影响MTase的酶活性但对复合物没有影响。形成或SAM结合27,29。因此,我们不能排除除了在MTase的稳定化中发挥作用外,Trm112还可能通过变构调节30在MTase的底物结合和活性调节中发挥作用的可能性。
生化和诱变分析
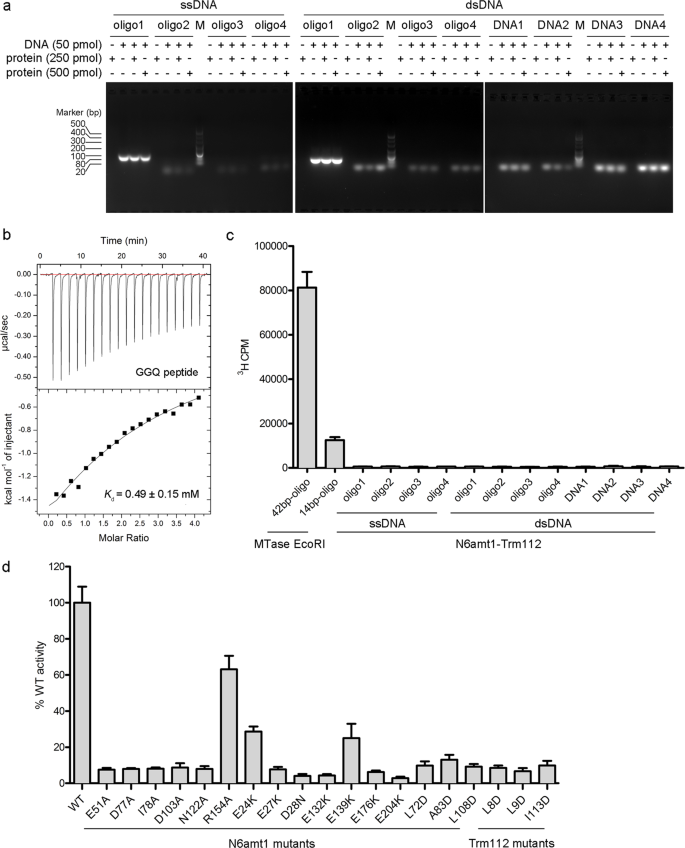
Previous studies on DNA 6mA modification in eukaryotes showed that the abundance and distribution of 6mA on the genomes and the identified 6mA enriched sequence motifs vary substantially in different eukaryotes. For instance, 6mA is enriched at the transcription start sites and an ApT dinucleotide motif was suggested to be the consensus sequence in C. reinhardtii3. However, 6mA is broadly and evenly distributed on the genome and two sequence motifs (AGAA and GAGG) were reported to associate with the presence of 6mA modification in C. elegans4. Furthermore, 6mA is shown to be highly enriched in exon regions in human genome and the [G/C]AGG[C/T] motif was identified to be significantly associated with 6mA modification10. Thus, to investigate whether the N6amt1–Trm112 complex could bind DNA, we first synthesized four DNA oligos according to those reported 6mA-enriched sequence motifs and four other DNA oligos used by Xiao et al. in the examination of the DNA 6mA modification activity of N6amt110, and performed in vitro electrophoretic mobility shift assay (EMSA). Our results show that N6amt1–Trm112 has no detectable binding with the tested dsDNAs or ssDNAs with different lengths and sequences, indicating that N6amt1–Trm112 cannot bind DNA in vitro (Fig. 3a).
针对不同ssDNA和dsDNA的N6amt1-Trm112 的 EMSA分析。Oligos1-4是Xiao 等人使用的四种DNA寡核苷酸。在检查N6amt1的DNA 6mA修饰活性10。其他四种寡核苷酸如下:DNA1,CTTTTTAGAAGCACA; DNA2,AATGAGAAGTTTATC; DNA3,AAAATCGAGGTTCCC; DNA4,ACTGACCATGGATCG。b ITC分析N6amt1-Trm112对eRF1的15残基GGQ肽的结合亲和力。在滴定之前,将蛋白质复合物与SAH一起温育。显示解离常数(K d)。CN6amt1-Trm112对不同长度和序列的dsDNA和ssDNA的酶活性测定。MTase EcoRI用作阳性对照。所有实验一式三份进行,误差棒代表平均值的标准误差。d N6amt1和N6amt1-Trm112界面活性位点关键残基突变对N6amt1-Trm112对人eRF1甲基化的酶活性的影响。野生型蛋白质复合物的平均3 H掺入测量(约2300cpm)定义为“100%WT活性”,并且相对于100%WT活性显示N6amt1-Trm112突变体的MTase活性。所有实验一式三份进行,误差棒代表平均值的标准误差
作为N6amt1报道用作用于ERF1特定谷氨酰胺转移酶23和能够催化与KKHGRGGQSALRFAR(此后命名为GGQ肽)的序列的人ERF1的Gln185为中心的15残基肽的甲基化31,我们再进行等温滴定量热法(ITC)实验检查N6amt1-Trm112和GGQ肽之间的结合亲和力。我们的结果表明蛋白质复合物具有结合GGQ肽的能力,尽管具有相对低的结合亲和力(0.49±0.15mM)(图3b))。这些结果与结构数据一致,表明N6amt1的潜在底物结合位点基本上带负电,这适合于富含碱性残基的底物肽的结合但不适合DNA的结合。将结构和生物化学数据结合在一起,N6amt1-Trm112复合物显示出对eRF1的GGQ肽的结合亲和力,但不显示对dsDNA或ssDNA的结合亲和力。
我们进一步进行了体外MTase活性测定以探索N6amt1的底物特异性。与体外结合测定结果一致,我们的活性测定结果显示N6amt1-Trm112复合物不能催化具有不同长度和序列的dsDNA或ssDNA寡核苷酸的甲基化(图3c)。作为阳性对照,MTase EcoRI可以高效催化DNA底物的甲基化(图3c)。另一方面,N6amt1-Trm112复合物可以有效地甲基化蛋白质底物eRF1(图3d)。
为了验证催化反应中N6amt1和N6amt1-Trm112界面活性位点关键残基的功能作用,我们进行了诱变和生化研究(图3d))。N6amt1的辅因子结合(Glu51,Asp77,Ile78,Asp103和Asn122)中涉及的任何残基的单突变导致对eRF1的活性几乎完全丧失,因为这些突变会影响SAM的结合。突变R154A导致对eRF1的活性损失约40%,表明Arg154在辅因子结合中起次要作用。在N6amt1的潜在底物结合位点处的大多数酸性残基(Glu27,Asp28,Glu132,Glu176和Glu204)的突变也消除了对eRF1的活性,表明这些残基在底物结合中起重要作用。此外,N6amt1潜在底物结合位点的Glu24和Glu139突变适度降低N6amt1的活性,表明这两个残基参与但不起基质结合中的关键残基的作用。此外,N6amt1-Trm112界面残基的单突变(N6amt1的Leu72,Ala83和Leu108; Trm112的Leu8,Leu9和Ile113)严重损害对eRF1的活性,表明蛋白质复合物的形成对其至关重要。活动。总之,我们的结构和生物化学数据证明N6amt1-Trm112能够结合并催化eRF1而不是DNA的甲基化,因此是真正的蛋白质MTase而不是DNA MTase。此外,伴侣蛋白Trm112对于N6amt1的稳定性是必需的,并且可能通过变构调节在N6amt1的底物结合和活性调节中起间接作用。N6amt1-Trm112界面处的残基的单突变(N6amt1的Leu72,Ala83和Leu108; Trm112的Leu8,Leu9和Ile113)严重损害对eRF1的活性,表明蛋白质复合物的形成对其活性是至关重要的。总之,我们的结构和生物化学数据证明N6amt1-Trm112能够结合并催化eRF1而不是DNA的甲基化,因此是真正的蛋白质MTase而不是DNA MTase。此外,伴侣蛋白Trm112对于N6amt1的稳定性是必需的,并且可能通过变构调节在N6amt1的底物结合和活性调节中起间接作用。N6amt1-Trm112界面处的残基的单突变(N6amt1的Leu72,Ala83和Leu108; Trm112的Leu8,Leu9和Ile113)严重损害对eRF1的活性,表明蛋白质复合物的形成对其活性是至关重要的。总之,我们的结构和生物化学数据证明N6amt1-Trm112能够结合并催化eRF1而不是DNA的甲基化,因此是真正的蛋白质MTase而不是DNA MTase。此外,伴侣蛋白Trm112对于N6amt1的稳定性是必需的,并且可能通过变构调节在N6amt1的底物结合和活性调节中起间接作用。总之,我们的结构和生物化学数据证明N6amt1-Trm112能够结合并催化eRF1而不是DNA的甲基化,因此是真正的蛋白质MTase而不是DNA MTase。此外,伴侣蛋白Trm112对于N6amt1的稳定性是必需的,并且可能通过变构调节在N6amt1的底物结合和活性调节中起间接作用。总之,我们的结构和生物化学数据证明N6amt1-Trm112能够结合并催化eRF1而不是DNA的甲基化,因此是真正的蛋白质MTase而不是DNA MTase。此外,伴侣蛋白Trm112对于N6amt1的稳定性是必需的,并且可能通过变构调节在N6amt1的底物结合和活性调节中起间接作用。
讨论
随着在一些真核生物的基因组DNA中发现6mA修饰,已经进行了许多努力来寻找负责建立和去除该修饰的蛋白质机器。DNA6毫安变形例的特定脱甲基酶已报道和由几组验证4,5,11 ; 然而,特定的DNA MTase仍然未知。N6amt1是否是哺乳动物中真正的N 6 -腺嘌呤DNA MTase仍然存在争议。在这项工作中,我们进行了人类N6amt1-Trm112复合物的结构研究,并进行了体外生化研究,以研究N6amt1的底物特异性。
大肠杆菌化酶HemK,这是远亲人类N6amt1,显示出催化多肽释放的甲基化因子RF1和RF2 19,20。还报道了N6amt1与Trm112形成异二聚体,并且在哺乳动物中作为eRF1的谷氨酰胺特异性MTase起作用23。进一步的研究表明,除了eRF1之外,N6amt1可以甲基化额外的蛋白质底物,并且其甲基化活性需要GQX 3 R基序31,这表明N6amt1的底物特异性相对较低。虽然N6amt1与大肠杆菌 HemK有很大关系,但超出MTase结构域的这些酶的结构特征却截然不同。除了MTase域,大肠杆菌化酶HemK包含与RF1基板(补充图:S广泛相互作用的大的N末端结构域4A)32。另外,大肠杆菌 HemK 的底物结合位点相对封闭,底物的Gln残基插入活性位点的窄通道中(补充图S4b)。这些结果表明大肠杆菌HemK可能具有相对高的底物特异性。相比之下,我们对N6amt1-Trm112复合物的结构研究表明,N6amt1的潜在底物结合位点相对平坦和开放,另外超出MTase结构域的辅助结构元件的尺寸相对较小并且由可能的环组成。经历构象变化以结合不同的底物(图1a和2d)。这些结果与N6amt1具有低底物特异性的功能数据一致。
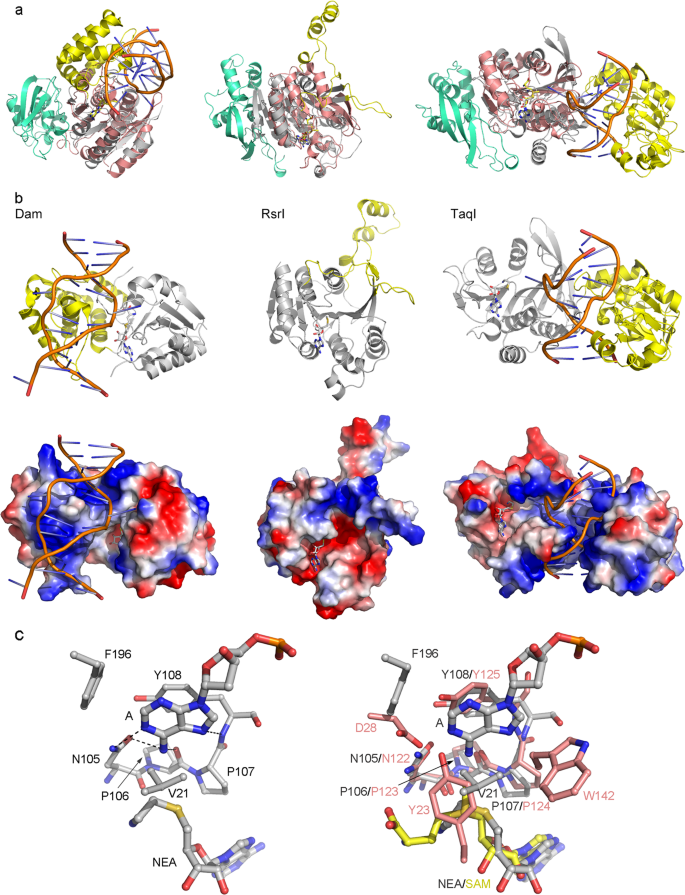
如今,关于N6amt1是否是哺乳动物中真正的N 6 -腺嘌呤DNA MTase的报道存在矛盾。N6amt1最初被提出作为一个SAM依赖性DNA转移酶,因为它具有的细菌n中的特性NPPY基序(基序IV)6 -腺嘌呤和N 4 -胞嘧啶DNA MTases 15,16。DNA MTases包括负责C 1 -C 5 MTases 5负责对于N -胞嘧啶甲基化和氨基MTases 6 -腺嘌呤和N 4 - 胞嘧啶甲基化。其中,所有C5 MTase由10个连续的保守基序(I-X)组成,以及C-末端附近的靶识别结构域(TRD)。与C5 MTase相似,氨基MTase具有一个TRD,但仅有十个保守基序中的九个(I-VIII和X)。氨基MTases可以被进一步划分为根据九个保守基序的命令三组(α,β,和γ),和所有三组含有DNA6毫安MTases 15,33。到目前为止,已报道了几种细菌DNA 6mA MTases的晶体结构。因此,我们进行了N6amt1-Trm112复合物与DNA 6mA MTases Dam,RsrI和TaqI的结构比较,分别代表氨基MTase的α,β和γ基团。与N6amt1一样,所有三种细菌DNA 6mA MTase都属于I类MTase家族28。这些酶的MTase结构域可以与N6amt1的MTase结构域良好叠加(对于140Cα和Dam,RMSD为3.0;对于50Cα和RsrI,RMSD为3.3;对于136Cα和TaqI,RMSD为2.2)(图。4A)。结构比较显示Dam,RsrI和TaqI的TRD位于MTase结构域附近,构成活性位点周围带正电荷的大部分表面的一部分,以促进DNA底物的结合(图4b)。结构分析显示构成I类MTases的核心MTase结构域的残基数平均为约150个氨基酸28。全长N6amt1仅含有214个氨基酸,辅助结构元件(L1环和插入结构域)仅包含约50个残基(图1a和补充图S 3b))。N6amt1的相对较小且主要带负电的辅助结构元件显然不利于酸性DNA底物与其活性位点的结合(图2)。
细菌DNA的结构分析6mA MTases显示基序X,I,II和III构成SAM结合区,基序IV-VIII构成催化区。在所有三组氨基MTase中,α和γ组的N末端以SAM结合区开始,而β组的N末端以催化区15开始。。对N6amt1的NPPY基序的序列位置和结构位置的分析表明,该酶类似于α和γ氨基MTase组比β氨基MTase组更多。当待甲基化的腺嘌呤碱基从双链DNA中翻出并插入TaqI-DNA结构中TaqI的活性位点时,我们比较了N6amt1的活性位点和TaqI的活性位点。比较显示,尽管周围残基的序列和结构相似性较低,但N6amt1和TaqI的NPPY基序可以很好地排列(图4c)。在TaqI-DNA结构中,腺嘌呤的亲核氮与Asn105的侧链羰基和Pro106的主链羰基形成两个氢键,并且嘌呤环通过与侧链的π-π堆积相互作用而稳定化。 Tyr108(图。4c)。在HemK-RF1复合物的结构中,RF1的谷氨酰胺残基插入HemK的活性位点,并以与DNA MTase TaqI类似的方式通过NPPY基序稳定(补充图4中的4d)。所提出的催化机制表明,氨基的基板和NPPY基序之间的氢键相互作用将偏振和从SAM激活氨基验收甲基的28,34。这表明N6amt1活性位点的NPPY基序可能具有催化DNA中腺嘌呤甲基化的能力。
一些以前的研究表明,N6amt1没有检测到的DNA转移酶的活性22,23,也没能得到一个DNA6毫安转移酶在胶质母细胞瘤干细胞11。一致地,我们的结构和生物化学数据表明N6amt1-Trm112复合物不具有结合DNA和催化DNA底物甲基化的能力。然而,先前的研究表明,向4个合成的DNA寡核苷酸重组Flag标记N6amt1展品6毫安修改活动10。当Flag标记的N6amt1从哺乳动物细胞中表达和纯化时,蛋白质样品可能含有一些内源DNA MTase,其负责DNA 6mA修饰。另一方面,如上所述,尽管N6amt1的推定的底物结合位点具有酸性,但N6amt1的活性位点的化学性质具有催化核苷酸碱基部分甲基化的潜力。由于N6amt1是一种相对较小的具有较小辅助结构元件的MTase,其伴侣蛋白Trm112参与N6amt1的稳定化,但不直接参与底物结合,我们不能排除可能存在可与N6amt1相互作用以掩盖活性位点周围的带负电表面并发挥细菌DNA MTase TRD功能以促进DNA底物结合的其他伴侣蛋白的可能性。 。以这种方式,N6amt1可以作为双功能性MTase,其可以催化蛋白质和DNA的甲基化,并且其活性可以由不同的伴侣蛋白质调节。
在准备我们的手稿期间,另一组发表了关于N6amt1-Trm112复合物35的结构和功能的研究。在他们的工作中,作者发现蛋白质复合物可以催化组蛋白H4K12的单甲基化,并解决了与SAH和H4K12me1肽复合的人N6amt1-Trm112的晶体结构。在该结构中,H4K12肽与N6amt1的酸性表面贴片结合,N6amt1被鉴定为该工作中潜在的底物结合位点35。他们的发现与我们的结果一致,即N6amt1的显着负电荷的底物结合位点不适合DNA结合。这些结果一起表明,N6amt1用作蛋白质转移酶可能甲基化组蛋白H4,ERF1,和另外的蛋白质底物31,35。
迄今为止,现有的结构和生化数据已经证明N6amt1对eRF1和H4都具有MTase活性。在与H4K12me1肽复合的N6amt1-Trm112的结构中,H4K12肽的甲基化赖氨酸的ε-胺原子与Asp28和Asn122的侧链羰基和Pro124的主链羰基形成三个氢键(补充图S 4e)。由于没有与eRF1复合的N6amt1-Trm112结构,我们通过叠加大肠杆菌将谷氨酰胺残基停靠在N6amt1的活性位点与N6amt1-Trm112的HemK2-RF1复合物,其显示RF1的谷氨酰胺残基的侧链氨基位于与H4K12的甲基化赖氨酸残基相邻的位点,表明eRF1的谷氨酰胺残基可被N6amt1识别。以类似的方式为H4K12(补充图S的赖氨酸残基4E)。此外,两个N6amt1-Trm112复合物的结构比较可知,形成N6amt1的活性位点的关键残基经历在所述底物结合(补充图:S没有大的构象变化4E)。这些结果表明,N6amt1的活性位点可以容纳和甲基化H4K12的赖氨酸和eRF1的谷氨酰胺。
材料和方法
克隆,表达和纯化
编码全长人N6amt1,Trm112和eRF1的基因通过PCR从293T细胞的cDNA扩增。对于N6amt1-Trm112复杂的表达中,N6amt1和Trm112基因克隆到pET-二重奏载体(Novagen),分别增加了一个他的MCS1和MCS2位点6处的N6amt1 N-末端标签。为了表达eRF1,将eRF1基因克隆到V28E载体中(来自中国科学技术大学生命科学学院的Tengchuan Jin博士的礼物)36,其中附有MBP标签和His 6标签。分别为eRF1的N末端和C末端。将构建的质粒转化到大肠杆菌中BL21(DE3)Condon Plus菌株。转化的细菌细胞在含有0.1mg / ml氨苄青霉素的LB培养基中于37℃生长至OD 600为0.6,然后用0.2mM IPTG在16℃诱导过夜。通过在补充有2mMβ-巯基乙醇和1mM苯甲基磺酰氟的缓冲液A(20mM Tris-HCl,pH7.5和100mM NaCl)中高压压碎,收集细胞,重新悬浮并在冰上裂解。通过离心沉淀细胞碎片,收集上清液用于蛋白质纯化。
N6amt1-Trm112复合物通过亲和层析纯化,使用Ni-NTA柱(Qiagen)和缓冲液A,缓冲液A分别补充有20mM咪唑和200mM咪唑作为洗涤缓冲液和洗脱缓冲液。使用用缓冲液B(20mM Tris-HCl,pH 7.5,100mM NaCl和1mM二硫苏糖醇)预平衡的Superdex G200 16/600柱(GE Healthcare),通过凝胶过滤进一步纯化蛋白质。MBP-eRF1-His 6的纯化除了在凝胶过滤中没有在缓冲液B中加入二硫苏糖醇外,蛋白质遵循相同的程序。Se-Met取代的N6amt1-Trm112复合物的表达和纯化遵循与天然蛋白质复合物相同的程序,除了细菌细胞在M9培养基中生长。使用QuikChange Site-Directed Mutagenisis试剂盒(Stratagene)产生含有点突变的N6amt1和Trm112突变体的构建体,并通过测序验证。突变体的表达和纯化与野生型蛋白质相同。通过SDS-PAGE分析,纯化的蛋白质具有高纯度(高于95%)。
结晶,数据收集和结构确定
将纯化的N6amt1-Trm112复合物在缓冲液B中浓缩至约16mg / ml。通过混合等体积的蛋白质溶液和储库溶液,使用悬滴蒸汽扩散法在16℃下进行结晶。天然蛋白质复合物的晶体在含有储库溶液(0.1M琥珀酸,pH7.0和15%PEG3350)的液滴中生长。硒-Met的衍生物蛋白复合物的晶体中含有储库溶液[1.6M的(NH滴中生长4)2 SO 4,0.1M MES,pH 6.5的,和10%1,4-二恶烷]。使用油对天然晶体进行冷冻保护,并使用补充有30%甘油的储库溶液对Se-Met衍生物晶体进行冷冻保护,然后闪蒸冷却成液体N 2。。在上海同步辐射装置的BL17U1和上海蛋白质科学国家设施的BL19U1中以100K收集衍射数据,并用HKL2000 37处理。衍射数据的统计数据总结在表1中。
使用Phenix 38中实施的单波长反常色散(SAD)方法解析Se-Met衍生物N6amt1-Trm112复合物的结构。使用衍生复合物结构作为搜索模型,通过分子置换(MR)方法解析天然N6amt1-Trm112复合物的结构。使用Coot 39进行模型构建,并使用Phenix 38和Refmac5 40进行结构细化。使用CCP4 41和PISA服务器42中的程序进行结构分析。使用Pymol(www.pymol.org)43生成结构图。结构细化的统计数据和最终结构模型的质量也总结在表1中。
eRF1和DNA的体外MTase活性测定
N6amt1用于ERF1的转移酶的活性测定在37℃下在50μl由4皮摩尔N6amt1-Trm112,各1μl的反应混合物中进行3 H-标记的SAM(珀金埃尔默)作为甲基供体,和40皮摩尔重组MBP- eRF1蛋白作为反应缓冲液中的底物(10mM Tris-HCl,pH 7.6,50mM KCl,10mM MgAc 2和14mMβ- 巯基乙醇)。1小时后,将反应混合物加载到Waterman P81滤纸上,将其风干,然后用10%三氯乙酸(TCA)洗涤三次并用丙酮洗涤10分钟。N6amt1的用于DNA的转移酶酶活性测定在25℃下在由20皮摩尔N6amt1-Trm112将50μl的反应混合物过夜进行,2.5μL 3H标记的SAM和250pmol DNA寡核苷酸作为反应缓冲液中的底物。将反应混合物加载到Waterman 3滤纸上,将其风干,然后用10%TCA洗涤三次并用乙醇洗涤20分钟。用液体闪烁定量掺入蛋白质或DNA底物中的放射性量。所有实验一式三份进行,误差棒代表平均值的标准误差。
等温滴定量热分析
使用等温滴定量热法(ITC)和ITC200微量热计(MicroCal)在20℃下测量N6amt1-Trm112和以Gln185为中心的eRF1的15残基肽(KKHGRGGQSALRFAR)之间的结合热力学参数。在滴定之前,将蛋白质复合物与S一起温育-Adenosyl-homocysteine(SAH)摩尔比为1:2。在该实验中,丢弃0.4μl肽溶液的初始注射以消除在平衡过程中滴定剂扩散穿过注射器尖端的影响,并且数据集包括20μl注射2μl2mM肽到样品池中250μl0.1mMN6amt1-Trm112。在实验中稀释的热量可以忽略不计。通过在MicroCal Origin软件7.0版中实施的非线性最小二乘法使用单个结合位点模型拟合积分滴定数据来确定结合常数和其他热力学参数。
电泳迁移率变动分析
进行EMSA以研究N6amt1-Trm112是否具有与不同长度和序列的几种dsDNA和ssDNA寡核苷酸的DNA结合能力。具体地,在含有10mM Tris-HCl(pH 7.5),50mM KCl和10mM MgAc 2的结合缓冲液中,20μl反应混合物由50pmol DNA组成。通过加入不同量的N6amt1-Trm112(250或500pmol)引发反应,然后在25℃下孵育2小时。将反应混合物加载到2.5%琼脂糖凝胶上,并在UV下用Gel Green(Biotium)染色显现。
入藏代码
天然和Se-Met衍生物N6amt1-Trm112复合物的晶体结构分别以登录号6KMR和6KMS保藏在蛋白质数据库中。