DAM簇的热响应遗传和表观遗传调控,控制桃花芽的休眠和低温需求
DAM簇的热响应遗传和表观遗传调控,控制桃花芽的休眠和低温需求
休眠是一种使多年生植物能够适应不利的季节性胁迫的适应措施。在冬季结冰是一个重大威胁温带植物在秋末进入休眠状态冬天到来之前,以避免冻害1,2。季节的环境因素主要是口述休眠发生,发展和释放,这已经在经历冬季休眠温带多年生植物了深入研究1,3,4。例如,光周期的缩短或温度的下降,或两者都在秋季引起桃(P. persica L. Batsch)的休眠,而温度下降是苹果(M. domestica)和梨的唯一因素(P. communis)。在所有情况下,环境因素引起的茎尖分生组织停止生长,形成花蕾进入生态休眠状态1,5。相反,由于顶端显性生长抑制而形成的侧生营养芽处于超休眠状态6。生态休眠和超休眠都是暂时的,可逆的,并且是秋天深秋5过渡到称为休眠气味的深度休眠状态的初始阶段。在夏天开始并形成的花蕾,类似地在秋天快要结束时进入内嗅7。异味是一种生理状态,除非暴露于低温(> 0-7.5°C)1下,否则在短时间的有利环境条件下不会轻易破坏或释放。该冷藏要求是强制性的,但在植物之间差异很大,这主要取决于来源和基因型8。同一树中不同芽之间或同一花中不同花器官的冷害要求也有所不同,这是由于休眠花芽相对于顶端叶芽7所需要的冷却时间更长,而雌花器官相比于雄花器官9所需要的冷却时间更长。因此,分生组织的生物学性质和发育轨迹也有助于冷却需求。
实际上,低温对于花卉发育至关重要。与被认为主要在细胞周期的G1期停滞并在寒冷时期保持静止的休眠植物芽10相反,休眠的桃花芽经历形态变化11,明显形成明显的原始孢子细胞和表皮,微孢子囊壁绒毡层和在花药12,13,并可见胚珠中心皮/雌蕊14,15,16。然而,这些花的形态变化很少发生在保持在室温或温暖温度下的完全休眠的花蕾中11。形态响应是,事实上,冷却阶段依赖性和主要的发展的事件,如在腕胚珠形成仅出现在接近冷却期间结束12,13,14,15,16,并且不足以冷却导致心皮的被捕胚珠形成之前或阶段发育9。显然,低温将休眠释放与花卉发育程序结合在一起,只有通过低温驱动的关键阶段(例如胚珠形成)才能使花卉芽从休眠中释放出来,或者能够在春季生长。
冷藏后立即加热的温度在协调花蕾发育和破裂中起重要作用。满足甚至需冷量之后,花芽仍然没有立即发展到开花,除非暴露一段温暖的温度,这种现象被称为“热和”的要求,这已在许多温带落叶果树被记录在案17,18,19。像制冷需求一样,热量需求是高度可遗传的,并且通常经过生态适应20。然而,温度等于或高于25℃经常地损害花器官发育21,22。寒冷和温暖的需求是相互关联和相互作用的,因此较长的寒冷时期导致较短的温暖时期的需求,这表明两种温度方案均以共同的基因或途径为目标。与此相反,暖要求,直接关系到芽的萌发和开花时间,并且不能由冷却被完全取代的10,18。
休眠的开始和退出,以及寒冷和温暖的需求似乎具有相似的遗传调控基础,这一开创性研究得到了关于桃长出(EVG)突变体的表征的开创性研究的证实,该突变体在茎尖分生组织和花蕾中均失去了休眠性23。基因组分析揭示6个串联重复高度保守的休眠相关MADS-box基因(DAM1-6)位于所述重量EVG轨迹,并确定了一个大缺失去除DAM1-4和沉默相邻DAM5-6突变内EVG轨迹24,因此提供了令人信服的证据,表明六种表达丧失DAM基因导致EVG表型。基因表达分析表明,在桃和其他李属品种中,夏末和初秋的顶端叶分生组织中DAM1,DAM2和DAM4上调,与它的生长停止和芽形成(生态休眠的一个阶段)相吻合,而DAM5和DAM6是在整个秋季增加,与从生态休眠到内生气味的过渡相吻合25。这些数据表明,这些DAM差异调节休眠的开始,这在转基因研究中得到了进一步的支持,在该研究中,DAM6的异位表达在杨树中促进生长停止,芽集和延长休眠期26。在杏仁(Prunus dulcis)中,PdDAM6在休眠释放期间,两个具有不同冷藏需求和开花时间的品种的转录本水平均持续下降27。
几条证据还支持DAM用作冷却温度的直接目标。首先,将负责冷藏需求的主要QTL特性定位到桃EVG基因座28。第二,DAM5和DAM6在冷却期或休眠释放11中被下调。第三,氢氰胺的应用促进了桃子休眠的中断,也下调了休眠植物人和花蕾中的DAM5和DAM6 29。第四,在DAM5和DAM6中都插入了转座子的品种需要较少的冷藏30。因此,特定DAM基因的下调或基因突变与休眠释放或降低的冷却需求相关。鉴于DAM s为同源的拟南芥短营养阶段,一个基因,其为转录抑制码特异性的目标,在平行于开花基因座Ç(FLC),花和器官鉴定的基因31,32,33,季节性振荡DAM可以直接协调花的发育进程以及休眠进入和释放:夏末秋季的表达增加会减缓或阻止花的发育进程(休眠进入),而冬季的表达降低会释放这种停滞(休眠释放)。
考虑到营养芽和花芽以及雄性和雌性器官之间的低温需求变化,桃中的六个DAM基因簇如何调节芽和器官特有的低温需求以及休眠开始和释放。另外,在相同的芽或组织中是否所有DAM都被相似或不同地调节尚不清楚。在拟南芥中,通过冷辅助内含子非编码RNA(COLDAIR)诱导组蛋白甲基化H3K27me3 34,冷直接靶向转录阻遏物FLC,该辅助RNA将H3K27me3沉积在FLC染色质35上。DAM是否与FLC共享相似或不同的表观遗传调控仍有待研究。在这项研究中,我们着手解决这些问题并了解DAM如何通过冷却和随之而来的温暖温度制度进行遗传和表观遗传调控。
结果
永生(EVG)突变桃的低温需求评估。
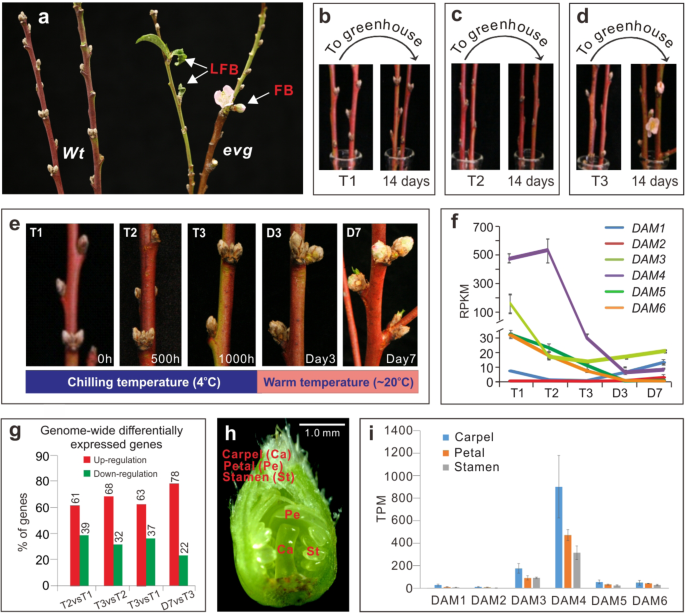
尽管桃EVG突变体在根尖分生组织和花蕾中失去了休眠开始,但其侧叶芽似乎保持了休眠和低温需求23,这引发了一个问题,即DAM是否全部参与了休眠和低温需求的调控芽。为了解决这个问题,我们通过将未经许可的生长条件下(〜20 °C)的EVG树上的切枝直接放置在允许的生长条件下(〜20 °C)来评估侧叶和花蕾的冷藏需求和休眠释放。同时,还包括来自Wt桃品种“ John Boy”的完全休眠的芽和切花芽的切枝作为对照。注意,evg树木仅形成花蕾和侧叶芽,但在收集样品时,顶端分生组织仍活跃。因此,在去除或不去除顶端分生组织的野生型和突变芽上进行了该实验。如果完全没有休眠限制,则EVG树上的花蕾和侧叶芽应继续生长或发育,并发展成芽折而不是停滞。图1a显示,所有Wt芽(例如,顶端和侧面的叶芽和花芽)在允许的条件下(即使在未进行1000 h冷藏的情况下进行预先处理)40天后仍被捕获(图1b–d)。但是从evg树继续断裂,生长和发育(右图,图1a)。我们的工作提供了令人信服的证据,表明DAM的缺失或其在EVG树中的表达不仅消除了根尖分生组织的休眠和寒冷需求,而且消除了侧叶和花芽。
一个在桃冷却无关的芽的萌发的确认EVG突变体。将在秋末从evg(右)和Wt(左)桃树收集的附有叶和花芽的芽直接置于正常生长条件下(〜20 °C)放置40天,无需进行冷冻处理。LFB侧生叶芽,FB花芽。b – d将完全休眠的Wt桃花芽在4°C下冷冻处理0 h(T1,b),500 h(T2,c)和1000 h(T3,d),然后恢复正常在温室(约20°C)下生长14天以测定芽断裂。ËT1,T2和T3冷藏花蕾以及温室(〜20°C)冷藏3天(D3)和7天(D7)的花蕾的形态。f DAM表达的RNA 序列分析,用RPKM绘制(每千个碱基对的读数/百万个作图的读数)。数据取三个生物学重复样本的平均值,为±SD。g每个阶段上调和下调的基因总数的百分比。h从休眠的T1-T3花蕾中解剖并收集花瓣(Pe),雄蕊(St)和心皮(Ca)组织。i RNA序列分析花器官特异性表达。数据取三个生物学重复样本的平均值,为±SD。每千千百万个映射读取的TPM成绩单
在休眠的花蕾中,低温冷淡地下调了六个DAM中的五个,并将DAM4鉴定为表达最丰富的一个
为了了解六种DAM在花蕾休眠释放过程中如何通过冷调节,我们分析了在经过低温条件(4°C)处理0,500后野生型品种“ John Boy”休眠花蕾中它们的表达和调控。和1000 h(分别为T1,T2和T3),然后转移至允许的生长条件(20°C)以诱导芽破裂(如图1b–d所示)。已经开发出这种冷却需求的体外测定方法,并已使用了几十年36,可以准确评估寒冷和随后的温暖温度对休眠释放或芽破裂的影响,而不会出现在野外条件下经常发生的干旱,冻结和/或短暂的温暖天气。将完全冷藏的T3芽(1000个冷藏小时(CH))转移到温室中允许的生长条件(20°C)后才开花(图1b–d)。形态上,冷的T3芽在温热处理后第3天(D3)略有膨大,并在第7天(D7)发育成完整的花瓣(图1e),表明1000 CH完全从休眠限制中释放了花芽。相反,T1和T2冷冻芽均未表现出明显的形态变化,也未出现芽断裂(图2)。1b,c,e)。因此,我们从处理过的芽的T1,T2,T3,D3和D7阶段(图1e)收集了处理过的芽的花组织(无芽鳞),用于RNA-seq,链特异性RNA-seq(ssRNA-seq),小RNA序列(sRNA-seq),全基因组亚硫酸氢盐测序(BS-seq)和ChIP-seq分析如下所述。
首先,我们发现,显著冷却下调五(DAM1,3,4,5,和6)的六个的DAM秒(FDR <0.05),而DAM2表达一致的低和接近检测限(图1F)。有趣的是,这五个DAM表现出特定于冷却阶段的下调模式。DAM5和DAM6从T1逐渐下降到T3,而DAM1和DAM3从T1急剧下降到T2,而DAM4从T2急剧下降到T3。DAM之间的相对表达水平也有很大差异s。DAM4在T2 的所有DAM中最高(〜512 RPKM),其次是T1 的DAM3(〜170 RPKM),T1的DAM5和DAM6(〜30 RPKM),而DAM1在T1 达到最低的水平(仅〜7 RPKM)。因此,DAM4的表达比DAM3高至少3倍,比DAM5和DAM6高 17倍,并且比DAM1高 70倍。同样,下调DAM4从T2在T3 512 RPKM至约30 RPKM,表示大约有17倍的降低,相对于减少的相对小的振幅最深刻的观察到的变化DAM1,3,5,和6转录物丰度。以下温暖条件(20℃),DAM4,5,和6中进一步下调,而DAM1和3保持不变或略微上调,表明温暖治疗强烈增强的冷却强加压制DAM4,5,和6。排除压制DAM的可能性s可能是由于冷胁迫施加的普遍存在的全基因组转录抑制所致,我们分析了在寒冷时期的全基因组差异表达基因(DEG)(表S 1)。超过60%的DEG通过从一个州到另一个州的冷却而上调(T2对T1,T3对T2,T3对T1和D7对T3)(图1g),证实了DAM的下调是生物学特异性的而不是结果是由寒冷压力引起的整体抑制所致。
DAM3和DAM4在心皮中优先表达
为了了解DAM表达是否可能是花器官特异的,我们分别从T1到T3花中分离了心皮,花瓣和雄蕊(图1h),并将它们合并起来进行转录组分析。与图1i中显示的全花数据一致,DAM4在三个花器官中表达最丰富,其次是DAM3,而DAM1,DAM5和DAM6的表达最低(图1i)。但是,DAM3和DAM4的表达水平在心皮组织中,它们分别比花瓣和雄蕊高出近两倍或三倍(图1i)。有趣的是,DAM1,5,和6,而在较低的水平表达,不得不在心皮相对较高表达。
DAM区产生的sRNA 对冷热处理的差异反应
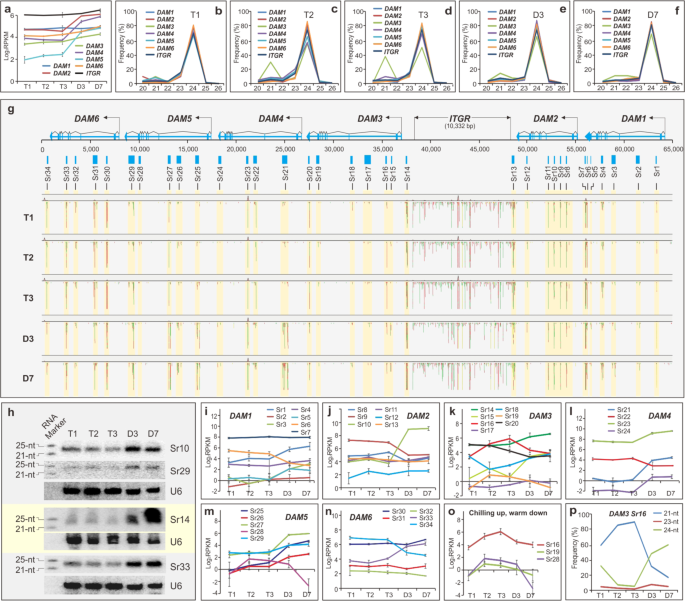
由于已知sRNA充当转录或转录后调节因子,因此我们接下来检查了低温和温暖的温度是否也调节DAM中 sRNA的产生。图2a中显示,的sRNA从所有六个产生DAM S以及10-kb的基因间区域(ITGR分隔)DAM1和2从DAM3,4,5,和6(图š 1)。在六个DAM和ITGR区域中,sRNA从T1到T3几乎没有变化,但除DAM1以外的所有其他方面均显着增加和ITGR从T3至D7,指示暖响应诱导。然后,我们分析了每个DAM中产生的sRNA的大小(20–26 nt)及其对治疗的反应。图2b-f显示,在所有分析区域中,24-nt sRNA是主要物种。有趣的是,DAM3区还产生了一个额外的21-nt sRNA物种,作为从T1诱导到T2的次要基团(图2b,c),并在T3进一步升高(图2d),然后在D3急剧下降(图2e)。 ,这与冷诱导的同源DAM3的下调相反(图1g)。查找DAM中产生单个sRNA的位置,我们将针对65 kb DAM基因组区域的sRNA读数作图。sRNA产生优先发生在基于共享表达模式分类的34个假定的sRNA产生区域或基因座(突出显示)中(图2g)。其中七个分别在DAM1和DAM3中,六个在DAM2中,五个在DAM5和DAM6中,四个在DAM4中。的锶基因座的大小而变化,范围从43到592-bp的但其中大多数是大于250-bp的(表比较短2)。有趣的是,仅三个Sr位点位于推定的启动子内(DAM3中的Sr14)或转录末端(DAM2中的Sr13和DAM6中的Sr34)。其余的31个位于转录区域内。这些31个位点中,25分别位于内含子内,其余6在内含子-外显子接点(或者居住Sr19在内含子7 -外显子8和SR20在内含子8 -外显子的9 DAM3,和SR24在内含子8外显子9 DAM4),或者在最后两个外显子(SR5,SR6,和SR7中的外显子9 DAM1,和Sr28在的外显子8DAM5)。要验证的Sr基因座独立地转录,我们进行RNA凝胶印迹分析和检测24-NT的sRNA生产中的代表性SR10,14,29,和33分别位点,但都没有从T1共享相同的表达模式到D7彼此之间或其同系的DAM(图2h),证实了一个独立的规定。
一个在六个的sRNA表达DAM秒。数据取三个生物学重复样本的平均值,为±SD。b – f sRNA种群(20–26 nt)和DAM s和T1(b),T2(c),T3(d),D3(e)和D7(f)的基因间区域(ITGR)的动态变化。g六个DAM区域中的sRNA定位图和sRNA产生区(Sr)/位置。具有类似sRNA表达活性的区域分为sRNA产生区域(Sr)/基因座和34 Sr突出显示,描绘和标记了基因座。红色表示反义笔录,绿色表示有义笔录。上面描述了带注释的六个DAM和ITGR。h RNA凝胶印迹。将约20 µg富集的小RNA样品与RNA标记一起溶解在16%的PAGE凝胶上,进行印迹,探测并用指示的p32标记的寡核苷酸和标记的RNA标记重新探测。sRNA标记的大小(21和25 nt)显示在左侧,U6作为对照。我 - p个体的sRNA表达锶在轨迹DAM1(我),DAM2(Ĵ),DAM3(k),DAM4(l),DAM5(m)和DAM6(n)。数据取三个生物学重复样本的平均值,为±SD。o低温使Sr基因座上调。p 21-nt sRNA主要在Sr16基因座表达
低温会优先诱导位于DAM3的Sr16的 21 nt sRNA ,而在不同DAM s中温暖上调编码24 nt sRNA的许多Sr位点。
我们检查了Sr基因座对冷热处理的反应。首先,所有Sr位点在热处理过程中均显示出不同的表达轨迹(图2i–n)。在34个Sr位点中,有21个对T3到D7的温暖温度响应,其中15个上调(Sr1,Sr3,Sr5,Sr10,Sr14,Sr15,Sr21,Sr23,Sr24,Sr25,Sr26,Sr27,Sr29,Sr30和Sr33)和6个下调(SR6,SR8,SR9,SR22,Sr32和Sr34),而八(的Sr 2,4,7,11,12,13,17和,31)没有响应的冷或暖的治疗,其中大多数位于DAM1和DAM2中。五个锶基因座(SR16,18,19,20,和28)响应了冷却处理:SR16,19,和28个上调,锶18和SR20下调。特别地,与其他相比,Sr16最丰富,并且显示出对冷的最强响应(图2o)。sRNA大小分析显示,Sr16是唯一编码21 nt sRNA的基因座(图2p)。因此,从T1到T3(图2b–d)在DAM3内检测到的冷诱导21-nt sRNA 完全由Sr16编码。
低温使DAM4中的 CHG和CHH甲基化增加,但温处理以序列上下文相关的方式差异调节DAM s 处的甲基化
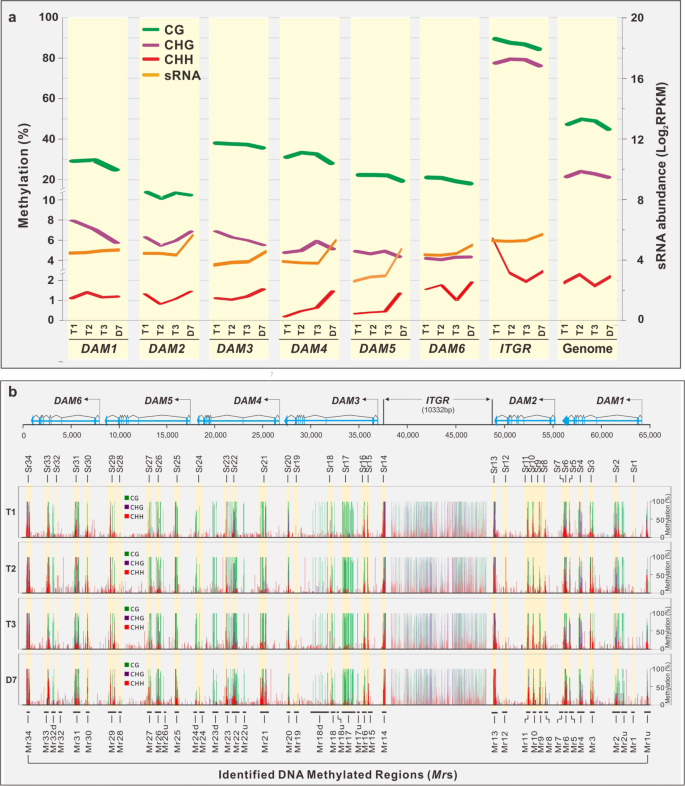
鉴于24-nt sRNA通过RNA依赖性DNA甲基化37引导DNA甲基化,我们进行了全基因组BS-seq的研究,以确定低温和温暖的温度是否调节DAM s 胞嘧啶的甲基化。图3a显示DAM的CG,CHG和CHH(其中H = A,T或C)序列上下文总体上是超甲基化的,但是甲基化轨迹对寒冷和温暖的温度的反应不同。分析的所有区域的CG甲基化在冷却期间(T1至T3)保持相对恒定,但在转变为温暖温度(T3至D7)后下降。除DAM4以外,所有地区的CHH甲基化在冷却过程中变化不大,其中观察到CHH甲基化增加。但是,温暖的温度使除DAM1以外的所有区域的CHH甲基化总体增加,这种增加在DAM4和DAM5中尤为明显。在这些相同的DAM中,该增加还与sRNA产生的增加相关(图3a)。显然,温暖的温度似乎反过来调节了CG和CHH甲基化。冷却温度对CHG甲基化的影响是多种多样的。它似乎增加了CHG甲基化DAM4但在减少DAM1和DAM3,并在变化小DAM2,5和6。显而易见的是,DAM4只是在CHH和CHG环境中冷却和在CHH环境中温暖而被甲基化的基因。
甲基化区域(Mr)与Sr区域的重叠
然后,我们调查了DNA甲基化是否与Sr基因位点重叠或与sRNA产生紧密相关。图3b显示甲基化不是随机分布在DAM中,而是聚集在大约44个区域(称为Mrs)中。大部分的先生轨迹重叠在锶除基因座SR1,19,和28,其中没有检测出甲基化。这些结果表明RDM在DAM中广泛存在。该先生区域是典型地比它们相应的较大的Sr基因座(表S = 2),与甲基化扩散到侧翼区域38一致。位于Sr重叠的Mr位点上游或下游的另外10个Mr区域(例如Mr1u,Mr2u,Mr17u,Mr18u,Mr18d,Mr22u,Mr23d,Mr24d,Mr26u和Mr32d),与任何Sr区域都没有重叠,表示siRNA依赖性甲基化的发生,这种现象在植物基因组中经常观察到。综上所述,先生的甲基化法规在寒冷和温暖条件下,不同序列背景下的不同区域是复杂多样的,但在许多条件下,在温暖条件下,CHH甲基化水平与sRNA丰度之间的整体相关性在许多Mr s中是显而易见的(图S 2)。
低温和温差诱导的H3K27me3及其传播
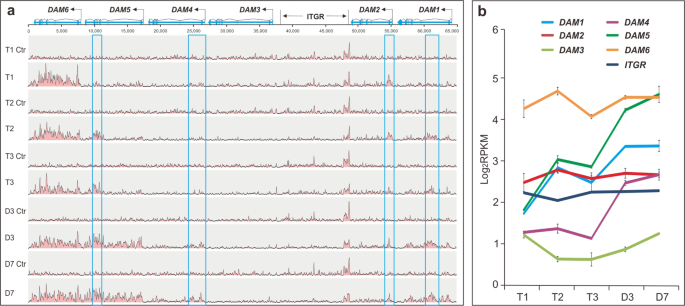
较早的研究表明,在休眠释放期间,某些DAM中由激冷引起的H3K27me3 39。我们进行了ChIP-seq,以了解在所有六个DAM中冷热温度如何调节H3K27me3 。该芯片起读取期间,在冷却期间富集DAM1,2,5,和6及的某些部分ITGR区域但其中不同富集的图案和位置(图4A)。在DAM1、2、5的内含子或外显子区域以及ITGR的一些区域中发生了小的局部富集,但在整个DAM6中是全基因范围的转录区。此外,所读取的富集改变作为富集峰的发生的时间范围在T2只出现以后在DAM1和5,但在T1向前为DAM2,6和ITGR,表明在读取富集DAM1和5是冷却依赖性(图4a)。在从T3的热情升温至D7,在读富集规律DAM2和DAM6沿ITGR仍然变化不大,但这些内DAM1和5经历了明显的变化,局部峰变大以覆盖整个转录区域,并且在DAM5中尤其稳健(下图,图4a)。有趣的是,尽管在冷却期间缺乏可见的富集峰,但DAM4在D3的转录起始区域附近也显示出一个主要峰,并在D7处向3'端扩散(图4a),表明温暖诱导并促进了扩散DAM4区域中H3K27me的数量。定量阅读富集分析进一步证实,寒冷和温暖的条件显着诱导和增强了DAM1和DAM5中的 H3K27me3 ,而温暖的情况则在DAM4中引起了区域(图4b)。温暖的温度也增强了DAM3和DAM6处的H3K27me3,尽管在读图谱中无法清楚地看出(图4a)。
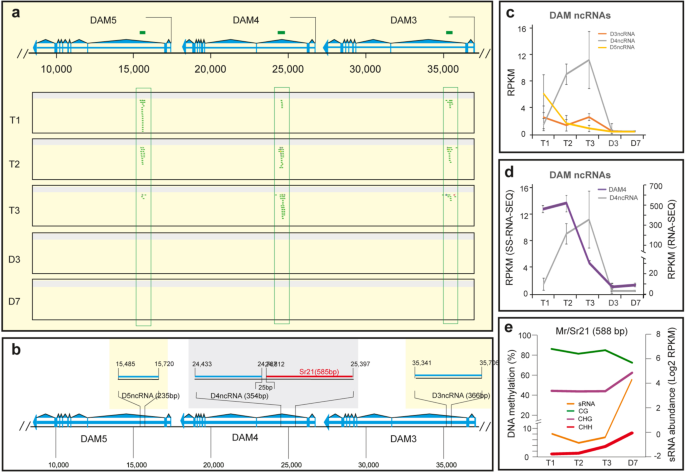
低温诱导DAM4区的非编码RNA(ncRNA)
进行链特异性RNA-seq以搜索对应于DAM的ncRNA。如图5a所示,丰富的有义读物特别地映射到DAM3-5的第二内含子,其分别大致对应于366-,354-和235-nt区域(图5b)。因此,我们将它们分别命名为推定的D3ncRNA,D4ncRNA和D5ncRNA。这三个ncRNA位于第二个内含子的相似位置(图5a,b),共有〜70%的序列同一性,但受冷差异调控(图5c)。D3ncRNAD5ncRNA保持不变或下调,而D4ncRNA从T1急剧上调至T2,并在T3达到最大表达水平(图5c)。然后,我们将D4ncRNA及其相关的DAM4的表达进行了比较,并揭示了在冷处理过程中强烈的逆相关性。在温暖的温度下,DAM4和D4ncRNA的表达均迅速下降,这表明在这两个阶段都可能起作用来抑制两者的共同机制。鉴于位于拟南芥FLC最大内含子中的可诱导低温诱导的COLDAIR被其自身的109-bp启动子转录35,我们检查了位于D4ncRNA区域上游的推定启动子区域的表观遗传变化。巧合的是,一个588 bp的Mr21 / Sr21基因座(如上所述)位于D4ncRNA编码区上游25 bp,并且有望与D4ncRNA启动子区重叠(图5b)。在温暖的温度下,Mr21 / Sr21基因座区域也与DAM4中的H3K27me3主峰重叠(图4a)。因此,我们专注于Mr21 / Sr21并发现24-NT的sRNA表达,CHH和CHG甲基化和的H3K27me3所有冷却期间保持在低或适度低的水平,但迅速从T3到上调D7(图4的a,b和图5e)。显然,推定的启动子或Mr21 / Sr21中 sRNA,DNA甲基化和H3K27me3的增加与温暖温度下D4ncRNA的强抑制共同相关。
针对六个DAM的链特异性RNA-seq读图的映射。与DAM转录方向相关的有义读取以绿色表示,反义以红色表示。有关DAM组织和位置的详细信息,请参见图S 1的图例。请注意,为清楚起见,删除了映射到外显子的读段。b ncRNA的位置和大小以及相关Mr / Sr基因座的示意图。c表达三个ncRNA。RPKM-每千位映射读取的每kb读取数。d从链特异性RNA-seq数据(SS-RNA-seq)中绘制D4ncRNA表达DAM4从常规RNA 序列数据中表达。È DNA甲基化和在表达的sRNA 先生 / SR21基因座
讨论区
花发育与响应季节温度的休眠开始和退出周期的耦合代表了一种关键的适应策略,即植物进化以应对温度胁迫以确保成功繁殖。在这里,我们表明寒冷和连续的温暖温度调节着多种表观遗传事件,这些事件可能协同使DAM在寒冷条件下迅速下调,并在生长期不可逆转地受到抑制,从而使花的发育速度得以成功进行,并且花蕾在适当的时间或季节。
确定DAM4是花蕾中的关键调控因子
早期的研究表明上调DAM1,2,和4枝条的生长停止期间顶端分生组织和芽开始,生态休眠阶段,和上调DAM5和6下落期间从生态休眠到休眠转变期间,和下调DAM5和DAM6在冬季25。总的来说,这些发现表明这些DAMs差异性调节了顶端叶芽中的休眠开始,发育和释放。在花蕾和侧芽中,DAM5和DAM6也显示由休眠释放期间冷却待下调11,26,29,这表明DAM5和DAM6参与在这些芽需冷量和休眠解除为好。在这里,我们的分析揭示了桃花芽中DAM的复杂调控,并表明六个DAM中的五个通过冷淡以不同的模式被下调(图1f)。此外,我们还鉴定出DAM4而不是DAM5或DAM6是表达最丰富的DAM基因,其表达量比其他4个高至少3-17倍DAM(图1f)。DAM4在心皮中优先表达(图1i),并且主要在寒冷时期的后期(图1f)被下调,据估计,DAM4在心皮胚珠的形成之前稍稍提前,胚珠是女性器官的关键阶段。开发14,15,16,40。鉴于总体关系DAM4与休眠退出和表达中记录了相应的独特和明显的后生事件DAM4,我们建议DAM4在花芽潜在密钥休眠调节器。DAM4通过冷却显示出17倍的表达降低,并保持在相当水平(〜30 RPKM)相当于在T1阶段DAM5或DAM6表现出的最高表达(〜30 RPKM)(图1f)。在温暖的温度下,DAM4持续下降并在D3以后达到最低水平,表明温暖进一步下调了DAM4。最近的QTL作图分析28还支持了DAM4在花芽休眠和芽断裂中的主要作用,该分析表明Pchgms40是位于DAM4的标记与分别位于D AM6的 Pchgms12或位于DAM5的Pchgms41相比,玉米与冷,热需求和芽断裂的性状紧密相关。综上所述,DAM4显然是桃花芽中冷,热需求和芽断裂的关键调节剂和性状变异性来源。
值得注意的是,DAM3还显示心皮,优先表达,并在较小的程度DAM1,5和6,表明这些DAM小号可能在调节休眠和萌芽表型以及发挥虽然次要角色的重要。五个DAM之间共享心皮优先表达将表明,由于其保守的序列和重复的性质,它们可能受到类似的调控。给定一个短间隔区(〜500-720个碱基)相邻之间存在DAM S(图š 1)中,密钥调控元件或增强要像在其中许多MADS框基因41,42,位于其中一个内含子中。替代地,六个DAM可以由单个增强剂共同调节,所述单个增强剂在远距离处激活或抑制DAM。该增强子可能(如果可能)位于DAM4区域,因为在EVG突变体中DAM1-4的缺失消除了DAM5和DAM6的表达24。如所预期的,如果是这样的情况下,DAM小号侧翼DAM4具有降低的表达正比于它们之间的距离,以DAM4(图1F,2克和S 1)。
低温驱动独特的表观遗传相互作用,从而为每个DAM定义独特的调节轨迹
在拟南芥,冷冻诱导的ncRNA和的H3K27me3沉默FLC 34,35。另一项有关杏仁的最新研究提供了与早期和晚期开花甲基化标记相关的编码区域的信息。还发现十个基因编码序列的甲基化状态与休眠释放过程43有关。在桃,冷冻诱导类似的后生反应,这之中的六个变化DAM S,用的H3K27me3诱导DAM1,5,和6,和SR16的sRNA在DAM3和D4ncRNA和CHG和CHH甲基化DAM4, 分别。这些DAM彼此之间或与拟南芥FLC没有相同的表观遗传调控,表明DAM特异的表观遗传调控。但是,所有DAM都普遍存在甲基化过高的现象(图3a,b),并且这种甲基化可能会为每个DAM添加额外的抑制层。因此,高甲基化与其他表观遗传元素之间的相互作用应有助于DAM之间表达丰度和模式的变化。
先前的工作表明,的H3K27me3在DAM1,4,5,和6中花芽野外条件下解除休眠期间被上调39,但由于没有明确定义的冷却和温暖的时段和休眠状态使得它很难区分冷却的作用连续的温暖温度。我们的工作表明,冷却和温暖作用明显不同:寒冷会诱导DAM1和5中的 H3K27me3局部化,而温暖会在整个基因范围内增强和传播它。在DAM4中,只有温暖而不是寒冷会诱导H3K27me3。但是,DAM6中的 H3K27me3发生冷却处理之前,并保持从冷却结束暖期间(图中的开始几乎不变4A,B),与早先的发现,即在的H3K27me3显然与DAM6休眠释放期间被上调39,44。因此,对H3H27me3的冷暖效应在DAM之间有所不同,在桃子品种之间也可能有所不同。有趣的是,的H3K27me3的存在,而不管丰度和阶段的,具有较低的表达(例如相关联的DAM1,5,和6),而没有将其与更高的表达(例如,相关DAM3和4)在冷却期间(图1f)。这可以解释为暗示H3K27me3对DAM具有总体抑制作用,或者较低表达的DAM可能易于产生H3K27me3。
激冷诱导COLDAIR在拟南芥FLC参与了蛋白质复合物沉积的H3K27me3招募35,但缺乏可检测的H3K27me3在DAM4在冷却期间指示该激冷诱导D4ncRNA功能有所变化DAM4。非编码RNA已显示出调节H3K9甲基化45,46,组蛋白脱酰,47和的蛋白质或转录因子募集48,49。D4ncRNA可能参与这些调节事件之一,以下调DAM4。Sr16编码的冷诱导21-nt sRNA在DAM3中的作用仍然是神秘的,它可能直接或间接抑制转录或转录后DAM3的表达。
温暖的处理增强了DAM的低温施加的表观遗传抑制
一个事实,即预热增强了冷却引起的,本地化的H3K27me3 DAM S(图4A,B)是具有对变暖增强冷却诱导的H3K27me3中的作用相一致FLC在春化拟南芥植物50,51。据信这种增强在生长季节中稳定并增强了FLC的抑制作用52。可以预期,桃花芽中H3K27me3的热诱导上调也可增强DAM基因的抑制。此外,我们还表明变暖与CHH甲基化的上调相对应,这在DAM4中尤为明显,5和6(图3a–d)。增加的甲基化与24-nt sRNA表达相关,表明变暖可能会激活DAM中的RdDM 。在同一DAM中,H3K27me3与CHH甲基化的并发应协同施加强于单个DAM的抑制作用,这由DAM4的连续下调以及温暖时期DAM5和6的稳定抑制来支持(图1f),因为所有三个DAM s的经受较强的H3K27me3和CHH甲基化相比DAM1,2和3(图3A和4a,b)。
DAM的热诱导和增强的表观遗传抑制在生物学上很重要
在冬季的拟南芥生态型,温暖的周期的几个星期(〜20℃)之后立即春化/冷却处理是用于建立和稳定春化/冷却效果的关键50。春化/冷藏的植物在立即置于30°C时失去开花的承诺,但在转移至30°C之前在20°C放置2周时仍保持开花。因此,春化/冷却后在20°C的温暖期对于稳定或增强“春化状态”至关重要。在桃花芽中也观察到类似现象21。完全冷藏的花蕾通常在20°C或低于20°C的温暖温度下正常发育,而在25°C或高于25°C的温度下异常发育。较高的温度通常会导致生殖器官特别是胚囊的停滞,并导致不良的坐果21。分子分析表明,在随后的温热处理(20°C或更低)下,春化植物中FLC染色质的冷诱导H3K27me3 会显着增强,但除非在30°C下暴露2周50才能显着降低。,51,它支持变暖(20°C)在寒冷诱导的春化状态的表观遗传强化中的作用。因此,桃花芽中温暖增强的H3K27me3和CHH甲基化应同样对DAM施加更强和稳定的抑制,但较高的温度(≥25°C)可以消除这种抑制,从而导致DAM在异位皮中表达损害正常胚囊和其他组织的形成或发展21。为此,表观遗传对温暖温度的反应的速率和效率可实现对DAM的强大且不可逆的抑制在不同的物种或品种中,s可能会有所不同,从而导致芽断裂或开花所需的不同温暖期或总热量。
材料和方法
休眠桃花芽的低温处理
桃品种“ John Boy”(Wt)和EVG的芽十月底,当芽中处于完全休眠状态时,从位于西弗吉尼亚州科尔尼斯维尔的USDA-ARS果园中生长的树木中收集树木。将收集的芽直接置于20°C进行芽破裂测定长达8周,或置于装有1/5水的容器中,并在4°C的黑暗生长室中于黑暗中冷冻0、500和1000 h (CH)。通过解剖和去除包围芽的芽鳞,在三个不同阶段(0(T1),500 CH(T2)和1000 CH(T3))收集花朵组织。冷冻处理后,将芽置于温室(约20°C)中以测定芽断裂,并收集在温室中保存3天(D3)和7天(D7)的芽的花蕾。在样品收集的每个时间点进行了三次重复。使用相同的组织分离RNA和DNA,以进行下面所述的RNA-seq,BS-seq,sRNA-seq和ChIP-seq分析。还从T1,T2和T3芽中解剖并分离了花瓣,心皮和雄蕊组织,并将同一组织汇集在一起,以分析花器官的特定花粉调节。DAM的
RNA-seq和链特异性RNA-seq数据分析
从花组织或器官组织中分离出具有三个生物学重复的总RNA样品。将约200 mg的花卉组织置于液氮中,并使用TriReagent(美国密苏里州圣路易斯的西格玛公司)提取,然后进行两轮苯酚-氯仿提取(50:50)。使用Illumina Hi-Seq将大约5μg的每个样品提交给BGI Americas合作公司(美国马萨诸塞州剑桥市),用于RNA-seq和ssRNA-seq。首先通过去除3'衔接子序列,通过CLC Genomic Workbench V.5,20(Qiagen,Hilden,德国)滤出rRNA和tRNA序列来处理RNA-seq读数。通过将生成的过滤后的读段直接映射到6个DAM准确标注的65kb区域,进行花器官特异性表达以及ncRNA和表达的分析。基于Peach基因组1.0 53(也参见图S 1)。将每个DAM或ncRNA区域中的读数计数分别归一化为每百万个图谱读数的转录本读数或每百万个图谱读数(RPKM)的外显子模型每千碱基的读数。根据CLC Bio(Qiagen,Hilden,Germany)提供的说明进行差异基因和转录本表达分析,并使用FDR 54校正多个测试的Raw P值。
芯片起和的H3K27me3分析DAM小号
在液氮下将约200–300 mg收获的桃花芽组织研磨成细粉,如先前所述进行细胞核分离和ChIP-seq 55。分离的染色质通过微球菌核酸酶(NEB,伊普斯威奇,马萨诸塞州,美国)消化。一半的核小体直接用于文库构建并测序作为阴性对照,而另一半则使用抗H3K27me3的抗体(Millipore 07-449)进行免疫沉淀。大约0.5到1 µg的回收的免疫沉淀DNA样品被送至康奈尔威尔医学院的核心机构,用于在HiSeq2000平台上进行文库构建和测序。每个样品进行两次重复。使用领结对ChIP-seq读数进行映射,并使用MACS2和CLC平台(Qiagen,Hilden,德国)进行峰调用。如图4b所示,对每千碱基对的每百万个映射读段(RPKM)的读段进行了计算和统计分析。
RNA凝胶印迹
如Zhu等[ 56]先前所述进行RNA印迹分析。简而言之,使用TriReagent(美国密苏里州圣路易斯的西格玛公司)从桃花芽中提取总RNA,然后进行两轮苯酚-氯仿提取(50:50)。使用mirVana miRNA分离试剂盒(AM1560,Thermo Fisher Scientific)进一步富集sRNA,并将约25 µg富集的sRNA印迹在尼龙膜滤膜上,然后对其进行探测并用32 P标记的Sr特异性探针重新探测。与的sRNA标记探针的探针一起标记有γ 32使用T4多核苷酸激酶(NEB,贝弗利,MA,USA)P-ATP。
小RNA测序和读取比对
如上所述,从总RNA中分离并富集小RNA,并如上所述连接至5'RNA衔接子和3'RNA衔接子57。将连接产物RT-PCR扩增并凝胶纯化,然后在Illumina HiSeq 2000平台上测序。对三个生物学重复进行测序。首先从原始sRNA读数中除去衔接子序列。进一步处理所得的sRNA序列,以去除那些包含低复杂度和t / rRNA序列且长度<15 bp或> 29 bp的sRNA序列。将其余的高质量sRNA读段与桃子基因组1.0进行比对,将65 kb DAM序列完美匹配,并将基因组中具有多个比对的读段排除在进一步分析之外。将每个sRNA的原始读取计数标准化为RPKM,并对DAM区域(Sr)期间,使用CLC Genomic Workbench V.5(Qiagen,Hilden,Germany)进行了温度依赖性的休眠释放和开花。
全基因组亚硫酸氢盐测序和数据处理
DNA样品3个重复,从使用DNeasy花组织中分离® Plant Mini试剂盒(Qiagen公司,德国希尔登),并提交给BGI美洲合作组织(剑桥,MA,USA)进行全基因组BS-Seq的,具有约30X基因组覆盖。对于未修饰的胞嘧啶,BS序列的错误转化率低于0.005%,与先前的数据58相当。为了使BS-seq读段与桃基因组对齐,首先将读段中的胞嘧啶碱基替换为胸腺嘧啶。然后使用Bowtie算法将转换后的读数分别与桃子基因组1.0的计算转换链对齐(一条链带C到T,另一条链带G到A),最多允许两个错配59。合并来自两条链的比对,并且每次读取仅保留最佳比对。分析中不包括多比对读取。然后将比对中的读取序列替换为原始的,未转化的序列59。最后,在比对的基础上,在全基因组范围内计算了每个胞嘧啶的甲基化水平,并沿着DAM区域显示了甲基化分布图(在CG,CHG和CHH上下文中)。
资料可用性
这项研究的所有原始数据都已提交到NCBI BioProject数据库(http://www.ncbi.nlm.nih.gov/bioproject/493230),登录号为PRJNA493230。
|
|